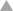OK. Jetzt wird's etwas komplizierter.....
Die Geräte an sich benötigen nicht nur einen READ?-Befehl. Wenn Du einen reibungslosen Ablauf möchtest, dann solltest Du Fehlerregister,... vor dem Absenden eines neuen Befehls leeren, das Gerät auf "bereit" schalten,.....
Die Befehle, die ich z.B. bei meinen Agilent-Geräten verwende siehst Du im Anhang.
Da habe ich Dir ein Bsp. für eine Gleichspannungsmessung gemacht. In der Realität habe ich da dann auch noch Widerstandsmessungen und AC-Messungen dabei, deren Befehle ich je nach Auswahl (per Cases) erzeuge.
Du solltest Dir auf jeden mal die Systembefehle anschauen (*CLS, *SRE 0,...).
Wenn Du die Befehle hintereinander schickst, dann musst Du aber aufpassen, dass ein "VISA Read" wirklich nur hinter einem Befehl kommt, der auch ein "?" enthält, also z.B. *IDN?, oder *READ?....
In meinem Bsp. siehst Du, dass ich zwischen den Befehlen ;: als Trennzeichen habe. Die verwende ich, um die Befehle nachher auseinanderzupflücken und auf ? zu überprüfen. Ist ein ? drin (True), wird ein Case mit VISA Read ausgeführt, ist kein ? drin, wird nur der VISA Write-Befehl ausgeführt.
Statt :; kannst Du auch irgendwas anderes verwenden. Es sollte halt nicht ein einzelnes Zeichen sein, dass in einem Befehl vorkommt.
Den Befehl kannst Du mit "Match Pattern" und einer Schleife mit Schieberegister zerlegen.
Gruß Markus
' schrieb:So ... ich hab an dem Kombinationsfeld gebastelt, da hat mir jmd hier: http://www.LabVIEWforum.de/index.php?showtopic=6924 weitergeholfen ...
Ich hab jetzt mal das wieder geändert, weil das mit dem CR auch nicht so recht wollte. Hab einfach mal das übernommen, was im LabVIEW <-> Serial.VI steht .. also das Texteingabefeld, und dann am ENDE CR/LF gemacht .. .das klappt jetzt ... aber nun gibts ne Statusmeldung, wenigstens keine Fehlermeldung mehr:
1073676294 Die übertragene Anzahl an Bytes entspricht der angeforderten Anzahl. Es sind möglicherweise weitere Daten verfügbar.
Und das Agilent gibt mir als Fehler: -410 Querry Interrupted.
Und nu bin ich mit meinem wenigen Latein am Ende ...